L’analyse la plus complète de la structure 3D du SRAS-CoV-2 à ce jour a révélé de nouvelles informations sur la façon dont le virus infecte les cellules humaines et se réplique.
Dirigés par le professeur Sean O’Donoghue, du Garvan Institute of Medical Research et du CSIRO’s Data61, les chercheurs ont compilé plus de 2000 structures différentes impliquant les 27 protéines du coronavirus. L’analyse a identifié des protéines virales qui « imitent » et « détournent » les protéines humaines – des tactiques qui permettent au virus de contourner les défenses cellulaires et de se répliquer.
Ces modèles structurels sont librement accessibles depuis le Ressource Aquaria-COVID, un site Web conçu par l’équipe pour aider la communauté des chercheurs à « zoomer » sur de nouvelles cibles potentielles sur le virus pour de futurs traitements ou vaccins, et à enquêter de manière cruciale sur de nouvelles variantes virales.

Crédit image : Institut Garvan
«Notre ressource contient un niveau de détail de la structure du SARS-CoV-2 qui n’est disponible nulle part ailleurs. Cela nous a donné un aperçu sans précédent de l’activité du virus », déclare le professeur O’Donoghue, premier auteur d’un article dans la revue Biologie des systèmes moléculaires détaillant les conclusions de l’équipe.
« Notre analyse a mis en évidence des mécanismes clés utilisés par le coronavirus ; ces mécanismes, à leur tour, peuvent guider le développement de nouvelles thérapies et vaccins. »
Informations structurelles
Pour mieux comprendre les processus biologiques, les chercheurs déterminent la forme 3D de protéines individuelles – les éléments constitutifs qui composent les cellules ou les virus.
« Les structures 3D des protéines nous fournissent des informations de résolution atomique sur la composition du SRAS-CoV-2 qui sont cruciales pour le développement de vaccins ou de traitements ciblant des parties distinctes du virus. Grâce à une récente focalisation de la recherche sur le SRAS-CoV-2, les scientifiques ont déterminé environ un millier de structures 3D des 27 protéines individuelles du virus, et près d’un millier de plus pour les protéines apparentées », explique le professeur O’Donoghue. “Cependant, jusqu’à présent, il n’y avait pas de moyen facile de rassembler toutes les données et de les analyser.”
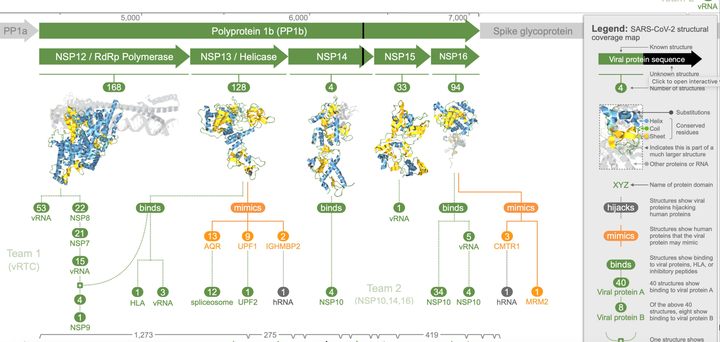
L’analyse de l’équipe a révélé trois protéines de coronavirus (NSP3, NSP13 et NSP16) qui « imitaient » les protéines humaines, ce qui, selon les chercheurs, permet au virus de mieux se cacher du système immunitaire humain et peut contribuer à la variation des résultats du COVID-19.
La modélisation a également révélé cinq protéines de coronavirus (NSP1, NSP3, glycoprotéine de pointe, protéine d’enveloppe et protéine ORF9b) que les chercheurs disent « détourner » ou perturber les processus dans les cellules humaines, aidant ainsi le virus à prendre le contrôle, à terminer son cycle de vie et à se propager à autres cellules.
«En outre, nous avons trouvé huit protéines de coronavirus qui s’auto-assemblent les unes avec les autres – l’analyse de leur assemblage a fourni de nouvelles informations sur la façon dont le virus réplique son génome. Cependant, après avoir pris en compte les chevauchements, cela laisse toujours 14 protéines qui, selon nous, jouent un rôle clé dans l’infection mais n’ont aucune preuve structurelle d’interaction avec d’autres protéines virales ou humaines », explique le professeur O’Donoghue.
« Pour rendre toutes ces informations et données plus accessibles aux chercheurs, nous avons conçu une nouvelle méthode de visualisation appelée carte de couverture structurelle. La carte met en évidence ce que nous savons sur le SRAS-CoV-2 et ce qu’il reste encore à découvrir – elle aide également les scientifiques à trouver et à utiliser des modèles 3D pour étudier des questions de recherche spécifiques.
Surveillance virale
L’analyse de l’équipe révèle des opportunités pour des recherches plus approfondies. « Une grande partie de la recherche sur les coronavirus à ce jour s’est concentrée sur la glycoprotéine de pointe, qui est la cible principale des vaccins actuels. Cette protéine continuera d’être une cible importante, mais il est également important que nous élargissions notre attention à d’autres cibles potentielles et comprenions mieux l’ensemble du cycle de vie viral », déclare le professeur O’Donoghue.

Crédit image : Institut Garvan
Il ajoute que la ressource Aquaria-COVID peut aider les chercheurs à étudier plus facilement en quoi les nouvelles variantes du coronavirus diffèrent – et surtout, comment elles peuvent être mieux ciblées avec des vaccins et des traitements.
«Plus le virus circule longtemps, plus il a de chances de muter et de former de nouvelles variantes telles que la souche Delta», explique le professeur O’Donoghue. «Notre ressource aidera les chercheurs à comprendre en quoi les nouvelles souches du virus diffèrent les unes des autres – une pièce du puzzle qui, nous l’espérons, aidera à faire face aux nouvelles variantes à mesure qu’elles émergent.»
La source: Institut Garvan