Les chercheurs ont franchi une étape essentielle pour comprendre la bataille microscopique entre nos cellules pulmonaires et le virus SARS-CoV-2 qui cause le COVID-19. Une étude dirigée par l’UC Berkeley a identifié des protéines spécifiques dans notre corps qui peuvent favoriser ou nous protéger contre les infections par le SRAS-CoV-2, ouvrant potentiellement la porte à de nouvelles thérapies antivirales.

Les chercheurs ont utilisé la technologie CRISPR pour tester l’impact de chaque gène humain sur les infections par le SRAS-CoV-2 dans les cellules humaines. Illustration par la co-auteure de l’étude Eleanor Wang, UC Berkeley
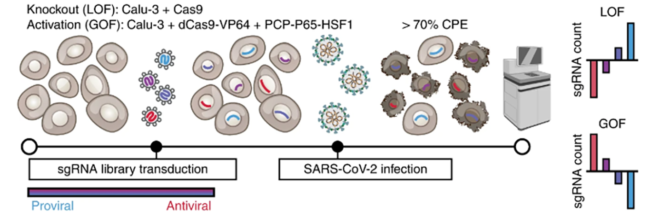
Dans l’étude, publiée dans Nature Genetics, les chercheurs ont utilisé la technologie CRISPR pour tester l’impact de chaque gène humain sur les infections par le SRAS-CoV-2 dans les cellules pulmonaires humaines. Leurs découvertes ont révélé de nouvelles voies sur lesquelles le virus s’appuie pour infecter les cellules et les voies antivirales qui aident à protéger contre l’infection virale. Notamment, ils ont montré que les mucines – le principal composant du mucus présent dans les poumons – semblent aider à empêcher le virus SARS-CoV-2 de pénétrer dans nos cellules.
“Nos données suggèrent que les mucines jouent un rôle clé dans la restriction de l’infection par le SRAS-CoV-2 en agissant comme une barrière contre les virus qui tentent d’accéder à nos cellules épithéliales pulmonaires”, a déclaré Scott Biering, co-auteur principal de l’étude et chercheur postdoctoral. dans le laboratoire d’Eva Harris à l’école de santé publique de l’UC Berkeley. “En outre, nos données suggèrent que les niveaux d’expression de la mucine dans les poumons d’un individu peuvent avoir un impact sur la progression de la maladie COVID-19.”
Patrick Hsu – cofondateur de l’Arc Institute et professeur adjoint de bio-ingénierie à Berkeley, Deb Faculty Fellow et Innovative Genomics Institute Investigator – est le chercheur principal de l’étude, qui a réuni des chercheurs de 10 institutions. Harris, professeur de santé publique à l’UC Berkeley, et Silvana Konermann, professeur adjoint à la Stanford University School of Medicine, étaient co-auteurs principaux de l’étude. D’autres collaborateurs ont contribué de l’Université de Stanford, de l’Université de Caroline du Nord à Chapel Hill, de la Yale School of Medicine et de l’Université Cornell. Ils couvraient des disciplines allant de l’immunologie, de la bio-ingénierie, de l’épidémiologie, de la biologie moléculaire et de la génétique.
Ensemble, les chercheurs essayaient de déterminer comment le virus SARS-CoV-2 pénètre dans les cellules humaines et se réplique si efficacement pendant la maladie. Ils voulaient également identifier des mécanismes de défense spécifiques dans les cellules humaines qui pourraient être capables de combattre l’infection, ce qui pourrait inspirer de nouvelles stratégies thérapeutiques.
Les chercheurs ont découvert que MUC1 et MUC4, des types de mucines présents dans les membranes des cellules pulmonaires, défendent les cellules pulmonaires contre l’infection. Cette découverte est importante car des études antérieures avaient suggéré qu’une accumulation de mucus pourrait être la raison pour laquelle certaines personnes sont tombées gravement malades avec COVID-19 – car le mucus peut rendre la respiration difficile pour les gens – et ont proposé d’utiliser des médicaments pour épuiser le mucus. Cette étude dirigée par Berkeley suggère qu’une telle stratégie pourrait interférer avec les mucines qui fournissent un mécanisme de défense précieux contre l’infection par le SRAS-CoV-2.
Pourtant, les mucines sont complexes et des recherches supplémentaires sont nécessaires pour les comprendre pleinement. Les chercheurs ont découvert que d’autres mucines – MUC5AC et MUC5B – qui sont sécrétées dans la muqueuse des poumons, ne font rien pour arrêter l’infection par le SRAS-CoV-2 ou peuvent même favoriser une infection virale.
Selon Biering, le type et la quantité de mucus que chaque personne produit peuvent entraîner des résultats différents pour l’infection par le SRAS-CoV-2 – et conduire à différentes stratégies de traitement.
« Quelqu’un qui produit beaucoup du bon type de mucus pourrait être très protégé. Mais quelqu’un qui produit beaucoup du mauvais type de mucus pourrait avoir plus de risque d’infection », a déclaré Biering. “Et quelqu’un qui produit très peu du bon type pourrait également être plus à risque.”
Il s’agit également de la première étude dans laquelle les chercheurs ont examiné comment le virus SARS-CoV-2 interagit avec les cellules pulmonaires humaines, marquant une avancée critique dans la recherche sur le COVID-19.
“Il est bien connu que l’infection virale peut être favorisée ou inhibée par nos propres protéines”, a déclaré Biering. “Mais c’est la première fois qu’une enquête systématique sur ces protéines de cellules hôtes est menée dans des cellules épithéliales pulmonaires humaines pour une infection par le SRAS-CoV-2.”
Des études antérieures sur l’infection par le SRAS-CoV-2 ont utilisé des types de cellules qui ne contiennent pas naturellement les récepteurs ou d’autres voies que le virus utilise pour infecter nos poumons. Pour cette étude, les chercheurs ont voulu utiliser un type de cellule plus pertinent, les cellules épithéliales humaines appelées Calu-3, trouvées sur la surface interne du poumon. Bien qu’il soit extrêmement difficile de travailler avec cette lignée cellulaire, les chercheurs ont réussi à acquérir une compréhension plus précise de la biologie impliquée dans les infections humaines par le SRAS-CoV-2.
“[Using Calu-3 cells] était un énorme progrès, étant donné qu’elles sont très représentatives des premières cellules que le virus contacte et infecte chez l’homme, et cela a révélé de nouvelles voies non vues dans d’autres lignées cellulaires », a déclaré la co-auteure principale Sylvia Sarnik, spécialiste adjointe du laboratoire de Hsu. au moment de l’étude. “Dans l’ensemble, cette étude est un pas en avant dans la compréhension des voies d’infection virale et ouvre la voie à la recherche vers de meilleurs traitements à l’avenir.”
Les chercheurs ont effectué un dépistage CRISPR de gain de fonction et de perte de fonction à l’échelle du génome pour éliminer ou surexprimer chaque gène du génome humain. Ensuite, ils ont mesuré l’impact de ces changements d’expression génique sur l’infection par le SRAS-CoV-2 dans les cellules épithéliales pulmonaires humaines. Il est important de noter que de nombreux gènes mis en évidence par cet écran n’ont pas encore été étudiés expérimentalement, fournissant un point de départ pour de futurs travaux.
“Vous pouvez exécuter ces expériences massivement parallèles de type” Hunger Games “sur des cellules humaines pour voir quels gènes vous pouvez modifier pour régler la capacité de nos cellules pulmonaires à survivre ou à se développer lorsque le virus les infecte”, a déclaré Hsu.
« Notre dépistage a identifié avec succès des centaines de gènes qui étaient importants pour la réplication du SARS-CoV-2 et des centaines de gènes qui pourraient restreindre le SARS-CoV-2 », a déclaré Biering. “Dans cette étude, nous avons choisi de nous concentrer sur la compréhension du rôle de ces protéines de mucine.”
Les chercheurs ont identifié les glycoprotéines de mucine comme des facteurs de restriction clés pour l’infection dans les cellules, à la fois dans des modèles murins et potentiellement chez l’homme.
Les chercheurs ont également étudié comment les mucines interagiraient avec d’autres virus respiratoires, notamment le virus de la grippe A, le virus parainfluenza humain, les coronavirus du rhume et le virus respiratoire syncytial. Comme pour le SRAS-CoV-2, les résultats étaient imprévisibles.
“Nous avons montré que les mucines sont largement antivirales, mais l’histoire est beaucoup plus compliquée que cela”, a déclaré Hsu. “En fait, dans certains virus, nous avons constaté que la surexpression de la mucine semblait en fait augmenter l’infectivité.”
Les travaux futurs seront essentiels pour mieux comprendre comment les virus interagissent avec les mucines, mais, pour l’instant, ces découvertes fournissent un point de départ important. “Cette étude nous a aidés à en savoir plus sur le virus et a ouvert de nouvelles voies de recherche pour étudier plus avant les cibles médicamenteuses”, a déclaré Sarnik.
La source: UC Berkeley